Analiza genów markerowych warunkujących wysoką plenność u owiec
Autorzy:
Grzegorz Smołucha, Agata Piestrzyńska-Kajtoch
Badania przeprowadzone na całym świecie na wysoko plennych rasach owiec (np. Booroola, Romney, Belcalre, Cambrige) wykazały, że za wysoką plenność tych ras mogą odpowiadać pojedyncze geny o dużym efekcie. Cechy reprodukcyjne zwykle charakteryzują się niską odziedziczalnością, a selekcja zwierząt oparta na ich fenotypie jest często powolna i nieskuteczna. W celu poprawy wskaźników cech reprodukcyjnych, skuteczniejsza wydaje się selekcja zwierząt hodowlanych oparta na ich genotypie. Liczne badania wykazały, że niektóre mutacje w genach BMPR-1B, BMP-15 i GDF-9 należących do nadrodziny transformujących czynników wzrostu-β (TGF-β) wpływają na poprawę płodności i plenności u tego gatunku zwierząt. Mutacje występujące w tych genach cechują się różnymi efektami oraz wzorcami dziedziczenia i mogą prowadzić do zwiększenia plenności i płodności jak również mogą powodować bezpłodność u osobników homozygotycznych, których jajniki nie rozwijają się prawidłowo.
Badania przeprowadzone w niniejszej pracy obejmowały identyfikację polimorfizmów w genach BMPR-1B (receptor białka morfogenetycznego kości 15), BMP-15 (białko morfogenetyczne kości 15)oraz GDF-9 (czynnik różnicowania wzrostu 9) u wybranych ras owiec objętych programem ochrony zasobów genetycznych. W tym celu pobrano materiał od 491 owiec ras: (55) Polska Owca Górska, (55) Merynos Polski ST, (64) Merynos Polski Barwny, (72) Świniarka, (35) Czarnogłówka, (55) Polska Owca Górska Barwna, Olkuska (50), (55) Wrzosówka i poddano go analizie za pomocą sekwencjonowania dideoksy Sangera oraz PCR-RFLP.
Gen BMPR-1B z mutacją, oznaczoną jako FecB (c.746 AàG), był pierwszym głównym genem plenności zidentyfikowanym u wysokoplennych owiec rasy Merynos. W ramach projektu BIOSTRATEG przeprowadzono badania oparte na analizie polimorfizmu genu BMPR-1B, z zastosowaniem enzymu restrykcyjnego AvaII (Wilson i in., 2001). Analiza otrzymanych wyników wykazała, że u wszystkich przebadanych zwierząt występował ten sam genotyp dziki (AA)i nie stwierdzono obecności mutacji w genie BMPR-1B, wykrytej pierwotnie u owiec rasy Merynos Booroola, charakteryzujących się wysoka plennością. Wyniki uzyskane metodą PCR-RFLP potwierdzono sekwencjonowaniem otrzymanych fragmentów, a otrzymane sekwencję porównano z dostępnymi w GenBanku (NC_019463.2). Wszystkie sekwencje były homologiczne do sekwencji występujących u owiec nie posiadających genu FecB. Otrzymane wyniki świadczą o braku segregacji tego allelu w polskiej populacji badanych ras owiec, nawet u wysokoplennej rasy owiec Olkuskich (Ryc.1).

Kolejnym genem poddanym analizie był gen BMP-15. Analizie poddano fragment genu BMP-15 (ekson1), kodujący peptyd sygnalny białka BMP-15 oraz fragment genu kodujący dojrzałe białko. Analizy otrzymanych wyników wykazały obecność delecji trzech nukleotydów c.28_30delCTT (aminokwas leucyna) (Ryc.2) w rejonie kodującym peptyd sygnalny białka BMP-15 u owiec rasy Wrzosówka.
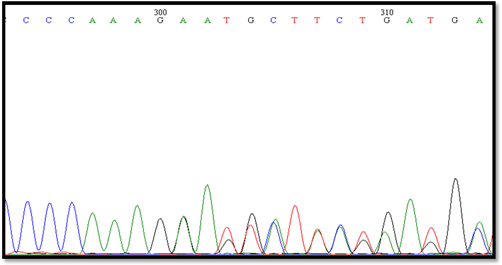
W przypadku pozostałych ras analiza otrzymanych chromatogramów nie wykazała obecności żadnej mutacji w eksonie I genu BMP-15. Hanrahan i in. (2004) zidentyfikowali polimorfizm w postaci delecji leucyny (bez efektów fenotypowych) sekwencji sygnalnej białka BMP-15 u owiec Cambrige i Belclare. W przypadku owiec rasy Wrzosówka niezbędny jest stały monitoring wielkości miotów oraz analiza rodowodowa w celu potwierdzenia czy zidentyfikowana delecja leucyny może być użyta jako marker plenności u tej rasy owiec a tym samym przynieść wymierne korzyści w hodowli tej rasy owiec. Dalsze analizy genu BMP-15 wykazały istnienie mutacji c.755T>C (g.6063) (rs55628000) w genie BMP-15 (Ryc.3.). Mutacja ta jest mutacją missensowną i powoduje zastąpienie leucyny (L) proliną (P) p.L252P w łańcuchu polipeptydowym białka (Ryc.4.).
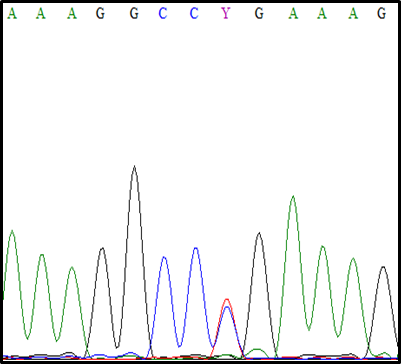
Zmiana ta nie skutkuje zmianą właściwości fizykochemicznych białka. Niepolarna, zasadowa leucyna zostaje zastąpiona niepolarną, zasadową proliną. Mutacja ta występuje przed sekwencją dojrzałego białka w pobliżu mutacji FecXG. We wszystkich analizowanych rasach owiec przeważał allel dziki TT. Mutację c.755T>C zidentyfikował Amini i in., (2018) u Irańskich owiec ras Afshari, Ghezel i Shal. Stwierdzili, że wykryta w tych rasach mutacja może być markerem zwiększonej plenności u tych ras owiec. W badanej populacji owiec rasa Polska Owca Górska charakteryzowała się największą ilością osobników będących nosicielami tej mutacji. Zidentyfikowana mutacja nie wpływała na zwiększenie plenności u tej rasy.
Kolejną mutacją zidentyfikowaną u badanych ras owiec za wyjątkiem owiec rasy Wrzosówka, Uhruska, Świniarka jest mutacja cicha niezmieniająca kodującego aminokwasu (Prolina) c.747T>C (Ryc.6) oznaczona przez Hanrahan i in.,(2004) jako mutacja B3 pierwotnie u owiec Cambrige i F700-Belclare. W przypadku ras świniarka i uhruska w analizowanych sekwencjach nie zidentyfikowano żadnego polimorfizmu.
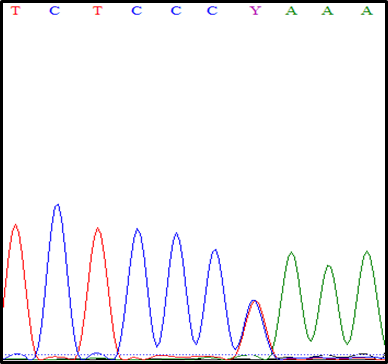
W przeanalizowanych sekwencjach genu BMP-15 otrzymanych w toku badań, u wszystkich badanych ras, nie odnotowano żadnej znanej mutacji sprawczej o udokumentowanym wpływie na plenność owiec tj. FecXG, FecXB, FecXI, FecXH, FecXL, FecXR, FecXGr za wyjątkiem owcy Olkuskiej, u której zidentyfikowano mutacje oznaczoną jako FecXO (c.1009A>C) (Ryc.5.) mającą potwierdzony wpływ na zwiększenie plenności w tej rasie owiec (Demars i in. 2013).

Wyniki wielu badań potwierdziły, że efekt mutacji FecXO daje inny efekt fenotypowy w porównaniu do dotychczasowych mutacji zidentyfikowanych w tym genie. SNP c.1009A>C zmienia kodujący aminokwas z asparaginy na histydynę, a wystąpienie mutacji w formie homozygotycznej CC nie skutkuje niepłodnością maciorek tak jak w przypadku pozostałych mutacji (wyjątek mutacja zidentyfikowana u owiec French Grivette FecXGr) a zwiększeniem plenności. Otrzymane wyniki po analizie genu BMP-15 świadczą o poligenicznym charakterze badanej cechy w populacji owiec objętych badaniami, za wyjątkiem owcy Olkuskiej.
Badania nad identyfikacją polimorfizmów SNP w genie GDF-9 przeprowadzono analogicznie jak dla genu BMP-15. Do badań użyto tą samą populację owiec jak w przypadku analizy genu BMP-15. Analizie poddano ekson I oraz II genu GDF-9, z czego region kodujący dojrzałe białko GDF-9 znajdował się na eksonie II. Porównanie sekwencji DNA u badanych osobników z sekwencją referencyjną zamieszczoną w GenBanku Acc. No. AF078545 skutkowało identyfikacją kliku mutacji punktowych. Mutacja c.477G>A oznaczona jako G3, c.471C>T oznaczoną jako G2, c.721G>A G4, c.978A>G G5, c.994G>A G6 (Ryc. 6.)

Wszystkie zidentyfikowane mutację zostały wcześniej opisane przez Hanrahan i in. (2004). U owiec rasy Cakiel podhalański zidentyfikowano mutacje SNP w pozycji c.1078T>C oraz g.1081T>C (Ryc.7.). Mutacja g.1081T>C nie powodująca zmiany w łańcuchu polipeptydowym białka natomiast mutacja g.1078T>C skutkuje zmianą kodującego aminokwasu z leucyny na prolinę.

W przypadku owiec rasy Olkuskiej zidentyfikowano trzy mutacje oznaczone G3, G5 oraz G6. U tej rasy owiec jak również przeważał genotyp dziki.
W pozostałych rasach owiec największą zmienność występowała u owiec: Polska Owca Górska Barwna, Polska Owca Górska, Cakiel podhalański. Może być to spowodowane wspólnym pochodzeniem tych grup owiec. Analizując otrzymane wyniki sekwencjonowania pod kątem wystąpienia mutacji sprawczych zwiększających plenność owiec tj. FecG, FecGH, FecGT, FecGE nie stwierdzono ich obecności w badanej populacji owiec. Zidentyfikowane mutacje tj., G2,G3, G4,G5, G6 nie wpływają na zwiększenie plenności owiec.
Literatura
- Amini, H.-R., Ajaki, A., Farahi, M., Heidari, M., Pirali, A., Forouzanfar, M., and Eghbalsaied, S.: The novel T755C mutation in BMP15 is associated with the litter size of Iranian Afshari, Ghezel, and Shal breeds, Arch. Anim. Breed., 61, 153-160, https://doi.org/10.5194/aab-61-153-2018, 2018.
- Demars J., Fabre S., Sarry J., Rossetti R., Gilbert H., Persani L., (2013), Genome-Wide Association Studies Identify Two Novel BMP15 Mutations Responsible for an Atypical Hyperprolificacy Phenotype in Sheep. PLoS Genetiv 9(4): e1003482.
- Galloway S.M., Gregan S.M., Wilson T., McNatty K.P., Juengel J.L., Ritvos O., Davis G.H., (2002), Bmp15 mutations and ovarian function. Molecular and Cellular Endocrinology, Volume 191, Issue 1, Pages 15–18
- Hanrahan J.P., Gregan S.M., Mulsant P., Mullen M., Davis G.H., Powell R., Galloway S.M., (2004), Mutations in the genes for oocyte-derived growth factors GDF9 and BMP15 are associated with both increased ovulation rate and sterility in Cambridge and Belclare sheep (Ovis aries). Biology of Reproduction, 70:900–9.
- Wilson T., Wu X.Y., Juengel J.L., Ross I.K., Lumsden J.M., Lord E.A., Dodds K.G., Walling G.A., McEwan J.C., O’Connell A.R., (2001), Highly prolific Booroola sheep have a mutation in the intracellular kinase domain of bone morphogenetic protein IB receptor (ALK-6) that is expressed in both oocytes and granulosa cells. Biology of Reproduction 64 1225–1235


