Charakterystyka struktury genetycznej polskich ras zachowawczych bydła, owiec, świń i koni w oparciu o mikromacierze SNP
Autorzy:
Artur Gurgul, Igor Jasielczuk, Tomasz Szmatoła, Ewelina Semik-Gurgul, Klaudia Pawlina-Tyszko
Jednym z celów badań była kompleksowa analiza struktury genetycznej populacji ras bydła, owiec, koni i świń uwzględnionych w programie ochrony ras zachowawczych oraz zwierząt kontrolnych ras produkcyjnych, z wykorzystaniem wysokowydajnych technik analiz genomu. Jako optymalne narzędzie dla celów genomiki populacji wybrano mikromacierze genotypujące firmy Illumina. Z wykorzystaniem tych macierzy badaniom poddano 796 owiec (OvineSNP50), 558 świń (PorcieSNP60), 911 sztuki bydła (BovineSNP50) oraz 576 koni (EquineSNP50).
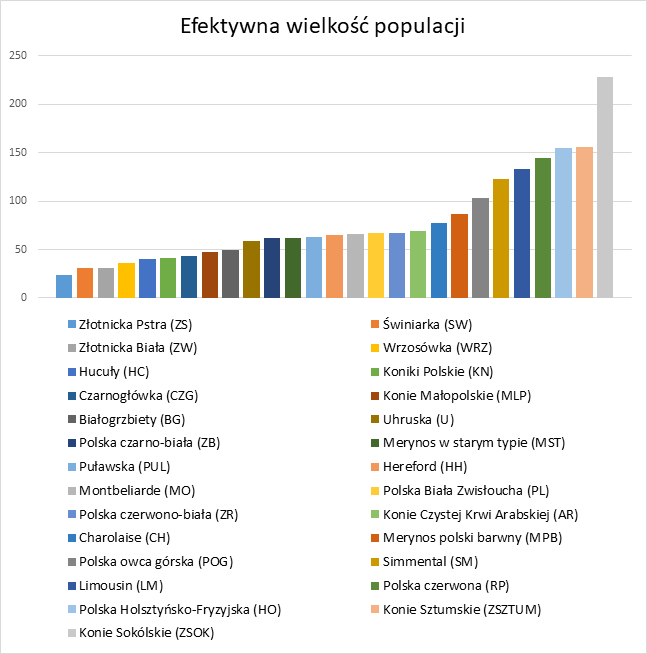
Wybrane dane odnośnie zróżnicowania i zmienności genetycznej analizowanych populacji przedstawiono poniżej (Ryc. 1-5) jako analizę głównych składowych zmienności (PCA).
Uwaga: Złap i przytrzymaj aby obracać model. Użyj rolki przewijania do powiększania i pomniejszania modelu.
W ramach badań określono także poziom zmienności i wsobności poszczególnych populacji w oparciu o genomy panel maklerów SNP. Szczegółowe dane odnośnie tych parametrów przedstawiono w pracy pt: „Ocena zmienności genetycznej oraz genetycznej podatności na wybrane jednostki chorobowe polskich ras zachowawczych oraz innych małych populacji zwierząt gospodarskich utrzymywanych w Polsce w oparciu o klasyczne i wielkoskalowe badania genetyczne”. Wybrane, poglądowe wyniki przedstawiono na rycinie 6 i 7.