Ocena genetycznej podatności polskich ras owiec na trzęsawkę – scrapie
Autorzy:
Agata Piestrzyńska-Kajtoch, Grzegorz Smołucha, Aldona Kawęcka
Trzęsawka (scrapie) to choroba neurodegeneracyjna owiec, kóz i muflonów, należąca do grupy pasażowalnych encefalopatii gąbczastych, która zawsze kończy się śmiercią na skutek całkowitej, fizycznej i umysłowej niewydolności organizmu. Trzęsawkę wywołuje akumulacja w centralnym układzie nerwowym (głównie w mózgu), tkance limfatycznej i innych organach, patogennego białka prionowego (PrPSc), tzw. białkowej cząstki zakaźnej (ang. proteinaceous infectious particle – prion). Agregacja i akumulacja prionów powoduje zamieranie komórek. Zamierające komórki nerwowe tworzą puste przestrzenie, przez co tkanka mózgu przypomina gąbkę [1, 4, 8, 15].
Trzęsawka ma długą historię. Pierwszy potwierdzony raport o tej chorobie owiec pochodzi z roku 1750 z Niemiec. Jednak publikacja brytyjska z 1772 roku wspomina przypadki scrapie z 1732 roku [4]. Objawy tej choroby są niespecyficzne, co znacząco utrudnia jej rozpoznanie (m.in. zmiany zachowania, miklonie, ataksja). Okres inkubacji choroby szacuje się na kilka m-cy do kilku lat [1, 2, 4, 5]. Trzęsawka owiec ma dwie postacie – klasyczną i atypową. Postać atypową odkryto dopiero w 1998 roku w Norwegii. Postać klasyczna uznawana jest za zakaźną (drogą wszczepienną, pokarmową i środowiskową), a atypowa za spontaniczną lub bardzo słabo przenoszącą się w warunkach naturalnych, pojawiającą się zwykle u starszych owiec i kóz [1, 4, 5, 18]. Różnice pomiędzy postaciami choroby są na poziomie molekularnym, w diagnostyce, a także w przypadku genetycznej podatności / oporności zwierząt [4, 18].
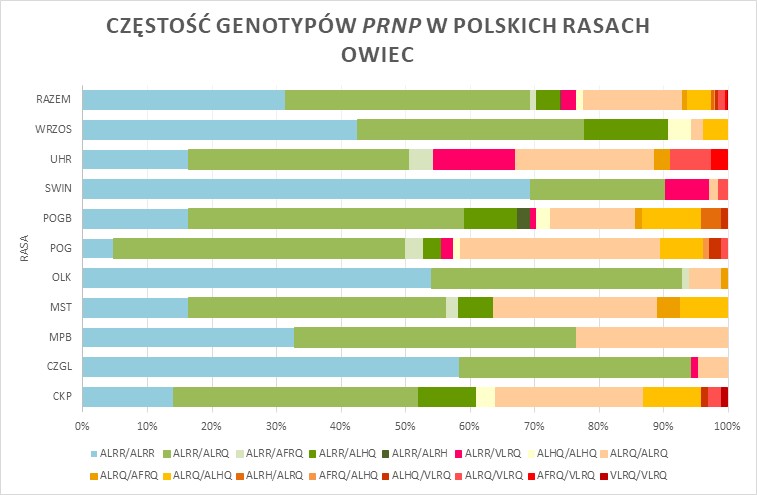
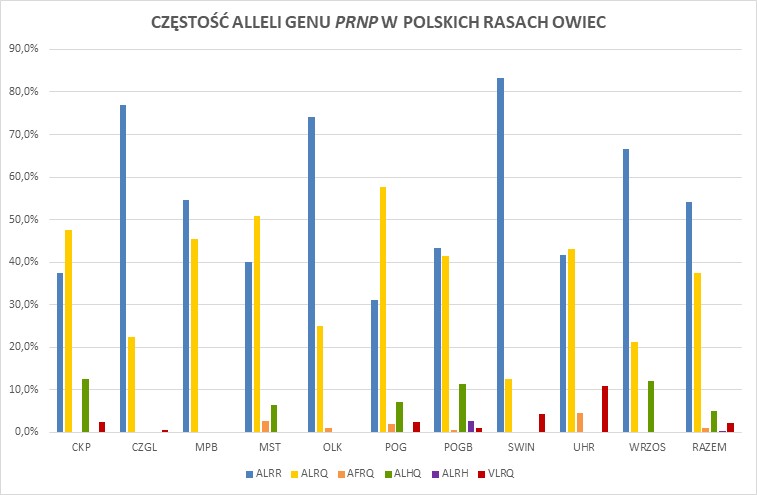
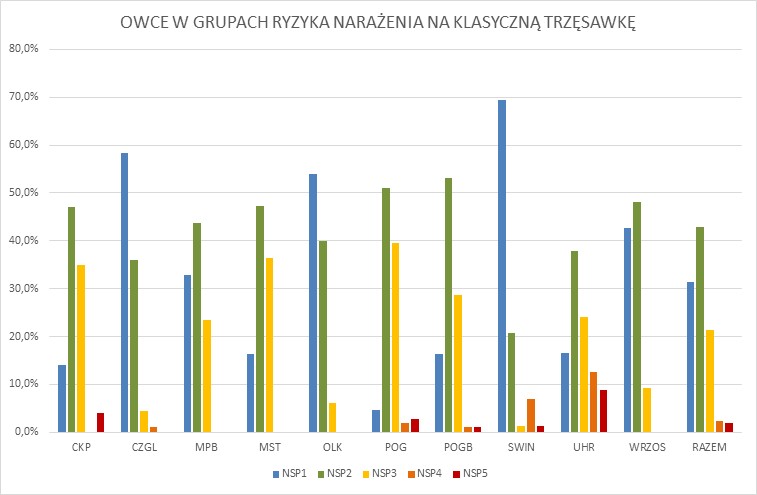
Stwierdzono, że polimorfizm kodującej części genu PRNP, którego wynikiem jest polimorfizm aminokwasów białka PrP (kodony 136, 141, 154 i 171), jest powiązany z występowaniem trzęsawki. W przypadku trzęsawki klasycznej, najwyższą oporność gwarantuje genotyp A136L141R154R171/ALRR, a najwyższą podatnością charakteryzują się osobniki posiadające genotyp VLRQ/VLRQ [1, 17]. Z trzęsawką atypową powiązano allele AFRQ i ALHQ, których częstość jest istotnie wyższa w grupie zwierząt z tą postacią choroby. Genotyp ALRR/ALRR nie chroni owiec przed zachorowaniem na atypową trzęsawkę [4, 5, 18]. European Food Safety Authority [3] klasyfikuje genotypy PRNP owiec na podstawie Great Britain’s National Scrapie Plan (NSP), przypisując je do pięciu grup ryzyka narażenia na tą postać trzęsawki owiec (NSP1: ARR/ARR; NSP 2: ARR/XXX; NSP 3: XXX/XXX; NSP 4: ARR/VRQ; NSP 5: VRQ/VRQ i XXX/VRQ; gdzie XXX=ARQ, ARH lub AHQ). Na całym świecie, także w Polsce, bada się genotypy genu PRNP [1, 7, 11-14, 16, 17, 19]. W wielu krajach opracowano programy selekcyjne oparte na eliminacji z populacji niekorzystnych wariantów genu PRNP i zwiększaniu częstości korzystnych wariantów tego genu.
W Polsce dotychczas (maj 2019) zdiagnozowano 87 przypadków trzęsawki owiec: 18 przypadków klasycznej trzęsawki i 69 przypadków trzęsawki atypowej. Wszystkie przypadki trzęsawki klasycznej zdiagnozowano u owiec pochodzących z importu.
Jednym z celów projektu był monitoring genotypów PRNP u owiec uwzględnionych w programie ochrony ras zachowawczych oraz zwierząt kontrolnych ras produkcyjnych. Niektóre dane przedstawiono na poniższych rycinach (1-3). Łącznie przebadano 817 owiec różnych ras, wykorzystując metodę sekwencjonowania i genotypowania SNP z sondami TaqMan MGB. Szczegółowe dane wraz z ich omówieniem przedstawiono w pracy naukowej pt. Ocena zmienności genetycznej oraz genetycznej podatności na wybrane jednostki chorobowe polskich ras zachowawczych oraz innych małych populacji zwierząt gospodarskich utrzymywanych w Polsce w oparciu o klasyczne i wielkoskalowe badania genetyczne.
Jeśli jesteś zainteresowany genotypowaniem trzęsawki owiec i/lub chcesz wykonać takie badania, skontaktuj się z nami: dr Agata Piestrzyńska-Kajtoch (e-mail: agata.kajtoch@izoo.krakow.pl , tel. 666 081 328), dr Grzegorz Smołucha (e-mail: grzegorz.smolucha@izoo.krakow.pl , tel. 666 081 204).
Literatura
Jeśli chcesz wiedzieć więcej o trzęsawce owiec, możesz przeczytać artykuły naukowe dotyczące tej tematyki. Poniżej znajdziesz wykaz piśmiennictwa wykorzystanego w tym opracowaniu. Wymienione artykuły nie wyczerpują zagadnienia, ale mogą stanowić podstawę do głębszego zrozumienia tej tematyki.
- Baylis M, Goldmann W. 2004. The genetics of scrapie in sheep and goats. Mol. Med., 4 (4): 385–396.
- Capucchio M. T., Guarda F., Pozzato N., Coppolino S., Caracappa S., Di Marco V. 2001. Clinical signs and diagnosis of scrapie in Italy: a comparative study in sheep and goats. Journal of Veterinary Medicine Series A, 48(1), 23-31. https://doi.org/10.1046/j.1439-0442.2001.00312.x
- EFSA (European Food Safety Authority), 2018. Scientific report on the European Union summary report on surveillance for the presence of transmissible spongiform encephalopathies (TSEs) in 2017. EFSA Journal 2018; 16( 11):5492, 64 pp. https://doi.org/10.2903/j.efsa.2018.5492
- Fast Ch., Groschup M.H. 2013. Classical and atypical scrapie in sheep and goats. In: W.-Q. Zou and P. Gambetti (eds), Prions and diseases, vol. 2, Animals, Humans and the Environment, Springer Science+Business Media, New York, 2: 15–44; https://doi.org/10.1007/978-1-4614-5338-3_2.
- Greelee J.J. 2019. Review: Update on classical and atypical scrapie in sheep and goats. Veterinary Pathology, 56(1), 6–16, doi: 1177/0300985818794247.
- Hagenaars T.J., Melchior M.B., Windig J.J., Bossers A., Davidse A., van Zijderveld F.G. 2018. Modelling of strategies for genetic control of scrapie in sheep: The importance of population structure. PLoS ONE 13(3): e0195009. https://doi.org/10.1371/journal.pone.0195009
- Kaczor U., Domon D., Martyniuk E., Murawski M. 2011. Polymorphism in the PRNP locus in prolific Olkuska sheep. Bulletin of the Veterinary Institute in Pulawy. 55. 3-7.
- Karmysheva V.Y., Pogodina V.V., Roikhel V.M. 2004. Cytopathological changes in human and animals brains in prion diseases. Behav. Physiol., 34 (5): 509–513.
- Konold, T., Phelan, L. 2014. Clinical examination protocol to detect atypical and classical scrapie in sheep. Journal of visualized experiments : JoVE, (83), e51101. https://doi.org/10.3791/51101
- Meling, S., Skovgaard, K., Bårdsen, K., Heegaard, P. M. H., Ulvund, M. J. 2018. Expression of selected genes isolated from whole blood, liver and obex in lambs with experimental classical scrapie and healthy controls, showing a systemic innate immune response at the clinical end-stage. BMC veterinary research, 14(1), 281.
- Niżnikowski R., Czub G., Świątek M., Głowacz K., Ślęzak M. 2014. Polimorfizm genu białka prionowego PrP u wrzosówki polskiej i owcy żelaźnieńskiej utrzymywanych w stadzie Doświadczalnej Fermy Owiec i Kóz SGGW w Żelaznej. Nauk. PTZ, 10 (2): 9–16.
- Niżnikowski R., Oprządek A., Świątek M., Czub G., Ślęzak M. 2016. Polymorphism of the PRNP gene in Polish Merino and old-type Polish Merino in flock with clinical status of atypical scrapie. Ann. Warsaw University of Life Sciences-SGGW, Anim. Sci., 55 (1): 99–107.
- Piestrzyńska-Kajtoch A, Gurgul A, Polak MP, Smołucha G, Zmudziński JF, Rejduch B., 2012. Characterization of PRNP and SPRN coding regions from atypical scrapie cases diagnosed in Poland. Mol Biol Rep. 39(3):2575-83; DOI: 10.1007/s11033-011-1010-0.
- Piestrzyńska-Kajtoch A, Kawęcka A., Smołucha G. 2017. Polimorfizm genu PRNP rodzimych ras owiec w aspekcie podatności na trzęsawkę (scrapie). Wiadomości Zootechniczne, R. LV, 5: 63–69.
- Prusiner S.B. 1998. Proc. Natl. Acad. Sci. USA, 95 (23): 13363–13383.
- Rejduch B., Knapik J., Piestrzyńska-Kajtoch A., Kozubska-Sobocińska A., Krupiński J., 2009. Frequency of genotypes in the PrP prion protein gene locus in the Polish sheep population. Acta Veterinaria Hungarica 57(1): 39-49.
- Tongue S.C., Wilesmith J.W., Cook C.J. 2004. Frequencies of prion protein (PrP) genotypes and distribution of ages in 15 scrapie-affected flocks in Great Britain. Vet. Rec., 154 (1): 9–16. Erratum in: Vet. Rec., 154 (4): 116.
- Tranulis M.A., Benestad S.L., Baron T., Kretzschmar H. 2011. Atypical prion diseases in humans and animals. Top. Curr. Chem., 305: 23–50.
- Wiśniewska E., Mroczkowski S. 2010. Prion protein (PrP) gene polymorphism and simulation study of breeding oriented to scrapie resistance in Polish Merino and Polish Mountain Sheep. Sci. Ann. Pol. Soc. Anim. Prod., 6: 115–123.


