Ocena podatności genetycznej polskich ras bydła na gąbczastą encefalopatię
Autorzy:
Igor Jasielczuk, Katarzyna Kaczanowska, Artur Gurgul
W ramach projektu dokonano oceny obciążenia populacji polskich ras zachowawczych bydła oraz rasy simentalskiej występowaniem niekorzystnych wariantów genetycznych warunkujących podatność na gąbczastą encefalopatię bydła (ang. Bovine Spongiform Encephalopathy, BSE). BSE to choroba neurologiczna, zaliczana do grupy pasażowalnych encefalopatii gąbczastych (Transmissible Spongiform Encephalopathy – TSE) wywoływana przez białkowe czynniki zakaźne (priony). Do podstawowych objawów BSE zalicza się zaburzenia neurologiczne i behawioralne, takie jak nadmierna reakcja na bodźce słuchowe i dotyk, nadpobudliwość, lękliwość, drżenia mięśni uszu i głowy, agresywność, brak koordynacji ruchowej, utrata wagi czy spadek wydajności mlecznej. BSE jest przewlekłą chorobą, a okres inkubacji jest bardzo zróżnicowany i wynosi od 2 do 8 lat (Cockcroft 2004). Objawy nasilają się powoli, jednak zawsze prowadzą do śmierci zwierzęcia. Dotychczasowe badania czynników genetycznych odpowiadających za podatność/odporność bydła na BSE doprowadziły do zidentyfikowania dwóch polimorfizmów insercji/delecji (indel) promotora i intronu 1 genu prionowego (PRNP), które wykazują powiązanie z występowaniem klasycznej odmiany choroby. Polimorfizmy te zlokalizowane są w intronie 1 (indel 12 pz) oraz w regionie promotorowym (indel 23 pz) (Hill i wsp. 2001; Zhao i wsp. 2015). Celem badań była analiza częstości polimorfizmów indel genu PRNP zlokalizowanych w intronie 1 (indel 12 pz) oraz w regionie promotorowym (indel 23 pz).
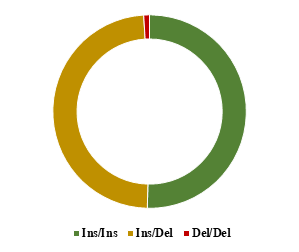
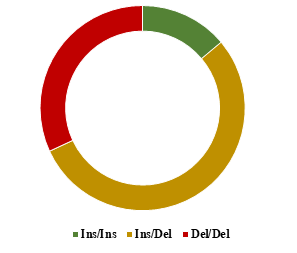
Poniżej przedstawiono graficzne zestawienie frekwencji genotypów oznaczonych w (A) locus polimorfizmu indel 23 pz promotora genu PRNP oraz (B) locus polimorfizmu indel 12 pz intronu 1 genu PRNP u przebadanych polskich ras zachowawczych bydła oraz rasy simentalskiej.
Kolorem zielonym zaznaczono udział potencjalnie protekcyjnego wobec BSE genotypu homozygotycznego pod względem insercji, kolorem czerwonym udział niekorzystnego genotypu homozygotycznego pod względem delecji, a kolorem żółtym genotyp heterozygotyczny.
Białogrzbieta
A
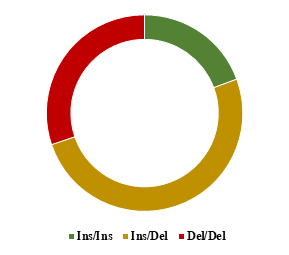
B

Polska czerwona
A

B
Polska czarno-biała
A
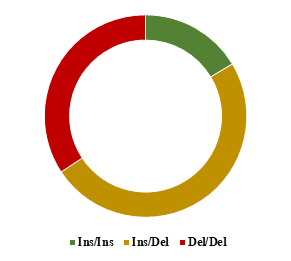
B

Polska czerwono-biała
A
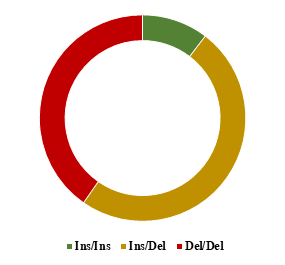
B
Simentalska
A

B
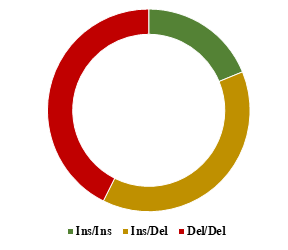
Spośród przebadanych ras bydła, zdecydowanie najniższym udziałem niepożądanych wariantów genu PRNP w obydwu polimorficznych loci charakteryzowała się rasa polska czerwona, co może świadczyć o mniejszej podatności osobników tej rasy na BSE. Największy udział niepożądanych wariantów genu PRNP w obydwu polimorficznych loci zaobserwowano u rasy simentalskiej, a w obrębie polskich ras zachowawczych u rasy polskiej czerwono-białej.
W ramach badań określono także udział frekwencji haplotypów obejmujących miejsce polimorficzne indel 23 pz oraz indel 12 pz genu PRNP u przebadanych polskich ras zachowawczych bydła oraz rasy simentalskiej.


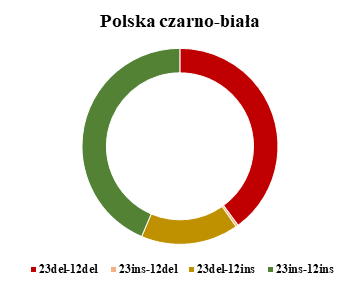
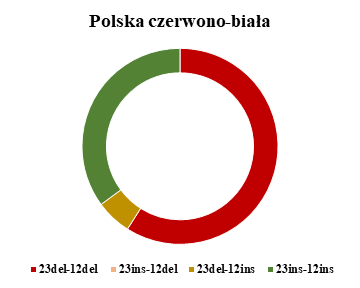
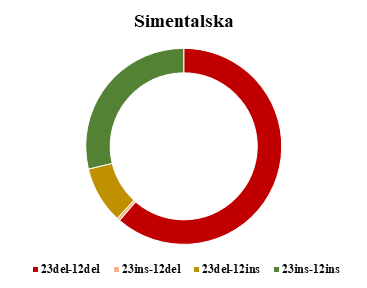
Analizując frekwencje haplotypów stwierdzono, że rasa polska czerwona charakteryzuje się najmniejszym udziałem niekorzystnego haplotypu 23 del – 12 del. Ogólnie u wszystkich przebadanych ras bydła stwierdzono relatywnie wysoki udział haplotypów 23 del – 12 del oraz 23 ins – 12 ins, co może świadczyć o zróżnicowanej podatności poszczególnych osobników wymienionych ras na BSE. Spośród badanych ras, najwyższy udział niepożądanego haplotypu 23 del – 12 del genu PRNP wykazano dla ras polskiej czerwono-białej i simentalskiej.
Szczegółowe dane odnośnie oceny obciążenia analizowanych populacji bydła występowaniem niekorzystnych wariantów genetycznych warunkujących podatność na BSE przedstawiono w pracy pt: „Ocena zmienności genetycznej oraz genetycznej podatności na wybrane jednostki chorobowe polskich ras zachowawczych oraz innych małych populacji zwierząt gospodarskich utrzymywanych w Polsce w oparciu o klasyczne i wielkoskalowe badania genetyczne”.
Literatura
- Cockcroft P.D. (2004) The similarity of the physical sign frequencies of bovine spongiform encephalopathy and selected differential diagnoses. Veterinary Journal 167, 175-80.
- Gurgul A. & Slota E. (2007) Effect of bovine PRNP gene polymorphisms on BSE susceptibility in cattle. Folia Biologica-Krakow 55, 81-6.
- Hills D., Comincini S., Schlaepfer J., Dolf G., Ferretti L. & Williams J.L. (2001) Complete genomic sequence of the bovine prion gene (PRNP) and polymorphism in its promoter region. Animal Genetics 32, 231-2.
- Zhao H., Du Y.L., Chen S.M., Qing L.L., Wang X.Y., Huang J.F., Wu D.D. & Zhang Y.P. (2015) The prion protein gene polymorphisms associated with bovine spongiform encephalopathy susceptibility differ significantly between cattle and buffalo. Infection Genetics and Evolution 36, 531-8.


